PHÁT HIỆN SỰ BẮT CẶP KHÔNG ĐẶC HIỆU CỦA MỒI 27F, 1527R KHI KHUẾCH ĐẠI GEN 16S rRNA TỪ DNA ĐA HỆ GEN VI KHUẨN TRONG MẪU PHÂN TRẺ EM
Viện Công nghệ sinh học, Viện Hàn lâm KH&CN Việt Nam
Số 18 Hoàng Quốc Việt, Cầu Giấy, Hà Nội
Số điện thoại: 0967060875; Email: huyendothi@ibt.ac.vn
Nội dung chính của bài viết
Tóm tắt (Abstract)
EFFECT OF ANNEALING TEMPERATURE ON NON-SPECIFIC AMPLIFICATION OF PRIMERS 27F, 1527R FROM METAGENOMIC DNA OF BACTERIA IN CHILDREN FECES
Primers 27F and 1527R are universal primers capable of amplifying nearly complete 16S rRNA gene of various bacteria in an environment for diversity analysis. In this study, the primers were used for amplification of multigene 16S rRNA of bacteria extracted from stool samples of a healthy child and a dirrheal child with unknown-pathogenic agent by three PCR programs: specific, tough-up and tough-down. The tough-up amplified DNA fragments of about 250 bp with high content from metagenomic DNA samples extracted by two methods from stool sample of diarrheal child. Sequences of the DNA fragments were 100% identical with each other, both contained 247 nucleotides. The DNA was the highest similarity (99.13%) with 22 genes encoding carbohydrate binding proteins (CBP) from B. longum. The investigating the binding sites of the two primers on the homologous genes showed that primers 27F, 1527R has a sequence of six nucleotides at the 3' terminal similarity to a sequence in the CBP gene, especially the 5/6 nucleotides at 3’ terminal of both primers were purines. Primer 27F was able to bind well but primer 1257R may not bind well to the 16S rRNA gene of B. longum. Thus, the low primer anealling temperature can help the primers binding well to the sequences which contains 6 nucleotides identical to the sequence at the 3' terminal of the primers.
Từ khóa (Keywords)
16S rRNA, PCR, non-specific amplification, primer 27F, primer 1257R, khuếch đại không đặc hiệu, mồi 27F, mồi 1257R
Chi tiết bài viết
1. ĐẶT VẤN ĐỀ
Để đánh giá đa dạng vi sinh trong môi trường sinh thái, các gen như gen mã hóa tiểu phần ribosome 16S (viết tắt là gen 16S rRNA), gen mã hóa dissimilatory sulfit reductase, nitrit reductase thường được sử dụng rộng rãi như gen đích trong các nghiên cứu, trong đó gen 16S rRNA được dùng phổ biến hơn cả [1]. Cặp mồi khuếch đại đa dạng (mồi phổ rộng) khuếch đại được đầy đủ nhất trình tự của gen 16S rRNA của nhiều loại vi khuẩn là cặp mồi 27F và 1527R [2-4]. Cặp mồi này được sử dụng phổ biến để đánh giá phân biệt các loài vi khuẩn có quan hệ di truyền gần gũi trong cùng một chi/loài và đánh giá đa dạng di truyền các loài trong cùng một chi khi kết hợp với một số phương pháp phân tích khác như điện di trên gel gradient biến tính (denaturing gradient gel electrophoresis - DGGE) vùng gen ngắn thuộc gen 16S rRNA, đa hình độ dài đoạn giới hạn (restriction fragment length polymorphism - RFLP) hay đa hình cấu hình sợi đơn (single strand conformation polymorphism - SSCP) [5] và giải trình tự gen. Ngoài ra, cặp mồi 27F và 1527R cũng được sử dụng để đánh giá đa dạng hệ vi khuẩn trong các môi trường sinh thái, điển hình như hệ vi khuẩn khử nitrat trong nước ngọt [6], đa dạng vi khuẩn trong nước biển [7], đất trồng ngô [8], trong bùn [9]... cho các mục đích khai thác nguồn vi sinh vật phục vụ phát triển nông nghiệp, công nghiệp. Tuy nhiên, việc đánh giá đa dạng vi sinh dựa trên kỹ thuật chỉ dấu phân tử (hay dấu vân tay phân tử - molecular fingerprinting techniques) phụ thuộc rất nhiều vào chương trình PCR, thành phần PCR và độ đa dạng DNA hệ gen của vi khuẩn trong mẫu [10]. Đối với mẫu từ hệ sinh thái có nhiều loại DNA khuôn từ các vi khuẩn khác nhau, việc sử dụng chương trình PCR có nhiệt độ gắn mồi (annealing temperature) đặc hiệu, mồi sẽ bắt cặp chủ yếu với các trình tự khớp hoàn toàn với mồi và sẽ bỏ sót lại những trình tự không khớp hoàn hảo. Do vậy, chương trình PCR đặc hiệu sẽ làm giảm độ đa dạng các trình tự được khuếch đại [10]. Mồi 27F được xem là mồi có mức độ bảo tồn cao nên không phát hiện thấy sự ảnh hưởng khi khuếch đại vi khuẩn trong mẫu nghiên cứu của Sipos và đồng tác giả [10] nhưng mồi 27F đã được báo cáo là không khuếch đại đa dạng được gen 16S rRNA của chi vi khuẩn Bifidobacterium và một số taxon khác có mặt trong mẫu phân [11]. Thông thường nhiệt độ bắt cặp của mồi với sợi khuôn để khuếch đại đặc hiệu trình tự đích được tính toán thấp hơn nhiệt độ Tm của mồi khoảng 3-5oC [12]. Do đó, nhiệt độ gắn mồi thích hợp cho khuếch đại đặc hiệu gen đích bằng cặp mồi 27F và 1527R là 52oC [2]. Nghiên cứu của Ishii và Fukui cho thấy khi tăng nhiệt độ mồi bắt cặp sợi khuôn, sản phẩm PCR được khuếch đại đặc hiệu hơn và ngược lại [13]. Chương trình PCR tough-up với nhiệt độ gắn mồi tăng dần ở một số chu kỳ đã giúp khuếch đại được các trình tự có số bản copy thấp có trong mẫu [14]. Trong khi đó tough-down PCR có nhiệt độ gắn mồi giảm dần để tăng khả năng gắn đặc hiệu cho mồi, làm giảm khuếch đại không đặc hiệu gen đích [15]. Với mục đích đánh giá đa dạng hệ vi khuẩn trong một môi trường sinh thái, việc khuếch đại được đa dạng các trình tự gen 16S RNA (bao gồm cả gen có độ tương đồng thấp với mồi, và gen từ vi khuẩn có tỷ lệ thấp trong mẫu) việc lựa chọn chương trình PCR cho khuếch đại gen là cần thiết.
Ở Việt Nam, tiêu chảy vẫn đang là 1 trong 10 bệnh phổ biến ở trẻ, trong đó có 2-20% trẻ tiến triển từ tiêu chảy cấp sang tiêu chảy kéo dài và có tới 47,39% trẻ không tìm được ra nguyên nhân gây bệnh [16]. Việc không tìm được ra nguyên nhân gây bệnh có ảnh hưởng lớn tới sự thành công của điều trị. Các nghiên cứu gần đây chỉ ra rằng, ở trẻ tiêu chảy không tìm ra tác nhân vi sinh gây bệnh là do rối loạn hệ vi sinh đường ruột, thiếu hụt lợi khuẩn bảo vệ niêm mạc ruột cũng như vi khuẩn giúp trao đổi chất cung cấp carbon mạch ngắn, vitamin,… cho cơ thể [17]. Sự mất cân bằng (rối loạn) hệ vi khuẩn trong mẫu phân thường được phát hiện thông qua phân tích gen 16S rRNA của nhóm vi khuẩn trội được khuếch đại từ đa hệ gen vi khuẩn [18]. Vì vậy, để nghiên cứu sự biến động của nhóm vi khuẩn trội trong mẫu phân bằng kỹ thuật dấu vân tay phân tử, chúng tôi đã tách chiết DNA đa hệ gen vi khuẩn từ hai mẫu phân: 1 của trẻ khỏe mạnh và 1 của trẻ tiêu chảy cấp (có triệu chứng viêm ruột, không xác định được tác nhân tiêu chảy bằng realtime-PCR dựa trên bộ kit Allplex™ GI-Bacteria(I) Assay (Seegene Inc)) bằng phương pháp phenol:chloroform của Sambrook và kit GenJET (GeneJET Genomic DNA purification kit (Thermo Scientific, USA)). DNA đa hệ gen này được sử dụng để làm khuôn cho thử nghiệm khuếch đại đa hệ gen 16S rRNA bằng các chương trình khác nhau. Tình cờ, chúng tôi đã phát hiện sự gắn không đặc hiệu của cặp mồi 27F và 1527R khuếch đại đoạn gen khác của Bifidobacterium khi chương trình PCR có sử dụng nhiệt độ gắn mồi thấp hơn nhiệt độ khuếch đại đặc hiệu. Các kết quả trong nghiên cứu này cung cấp các thông tin để các nhà khoa học có thể tham khảo trong việc thiết kế các đoạn mồi và thiết lập phương pháp PCR ứng dụng trong chẩn đoán hay đánh giá đa dạng.
2. VẬT LIỆU VÀ PHƯƠNG PHÁP NGHIÊN CỨU
2.1. Vật liệu
DNA đa hệ gen của vi khuẩn trong mẫu phân của trẻ khỏe mạnh (bé trai 12 tháng tuổi khỏe mạnh, không sử dụng kháng sinh và không bị mắc bệnh trong vòng 1 tháng trước nghiên cứu), bé gái 15 tháng tuổi (được chẩn đoán tiêu chảy viêm ruột nhưng không tìm được tác nhân gây bệnh âm tính với tác nhân vi khuẩn gây bệnh bằng realtime-PCR dựa trên bộ kit Allplex™ GI-Bacteria(I) Assay (Seegene Inc)) được tách chiết bằng phương pháp phenol:chloroform của Sambrook và kit GenJET (GeneJET Genomic DNA purification kit (Thermo Scientific, USA)) [19].
2.2. Phương pháp nghiên cứu
Phương pháp PCR khuếch đại đa gen 16S rRNA từ đa hệ gen vi khuẩn
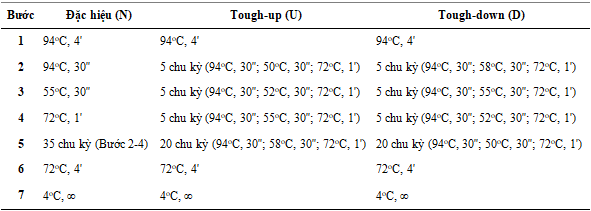
Đa gen 16S rRNA có kích thước khoảng 1,5 kb được khuếch đại từ DNA đa hệ gen bằng mồi 27F (5′-GAG TTT GAT CCT GGC TCAG-3′) và 1527R (5′-AGA AAG GAG GTG ATC CAGCC-3′). Tổng thể tích PCR (25 µl) gồm 2,5 µl đệm PCR 10x, 2,0 µl dNTP (2 mM), 1,0 µl mồi mỗi loại (10 µM), 5,0 µl DNA khuôn (2 ng/µl), 0,25 µl DreamTaq DNA polymerase (5 U/μl, Fermentas). Chương trình PCR được thể hiện trong Bảng 1. Sản phẩm PCR được kiểm tra bằng phương pháp điện di trên gel agarose 0,8%.
Bảng 1. Chương trình PCR khuếch đại gen 16S rRNA
Giải trình tự và phân tích trình tự
Vùng gen được khuếch đại với hàm lượng lớn, rõ nét trên điện di đồ, thể hiện là băng DNA trội trong mẫu có kích thước khoảng 250 bp được tinh sạch bằng MEGAquick-spin plus total fragment DNA purification kit (Intron, Hàn Quốc) và được giải trình tự trên hệ thống máy ABI 3100 bằng phương pháp Sanger. Gen được giải bằng mồi 27F và 1527R để thu được chiều dài đầy đủ của gen khuếch đại. Sắc ký đồ giải trình tự được quan sát bằng phần mềm Chromas và được so sánh tương đồng với các trình tự trên ngân hàng gen bằng công cụ BLASTN. Các trình tự mã hóa cho gen 16S rRNA chuẩn và các gen khác được lấy từ ngân hàng gen thông qua NCBI nucleotide. Các trình tự được so sánh tương đồng bằng công cụ CLUSTALW.
3. KẾT QUẢ VÀ THẢO LUẬN
3.1. Khuếch đại đa gen 16S rRNA, giải trình tự gen 250 bp
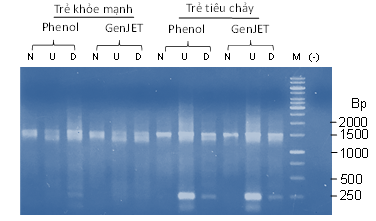
Hình 1. Điện di đồ phân tích sản phẩm PCR khuếch đại gen 16S rRNA bằng mồi 27F và 1527R từ đa hệ gen của vi khuẩn trong mẫu phân trẻ khỏe mạnh và trẻ tiêu chảy chưa rõ nguyên nhân
Phenol, GenJET: Đa hệ gen vi khuẩn trong phân được tách bằng phương pháp phenol:chloroform và bằng kit GenJET; (-): Đối chứng âm không chứa DNA khuôn; M: DNA chuẩn 1 kb (Fermentas); N, U, D: tương ứng với chương trình PCR đặc hiệu, tough-up, tough-down.
Kết quả (Hình 1) cho thấy đa gen 16S rRNA có kích thước điển hình 1,5 kb đã được khuếch đại thành công từ DNA đa hệ gen của vi khuẩn trong phân của trẻ khỏe mạnh và trẻ tiêu chảy được tách bằng cả hai phương pháp phenol:chloroform và kit GenJET. Tuy nhiên, phổ băng DNA được khuếch đại ở các chương trình PCR đã có sự khác nhau lớn. Đa gen 16S RNA chỉ được khuếch đại đặc hiệu khi PCR với chương trình đặc hiệu (N) từ tất cả bốn loại DNA khuôn (Hình 1). Các chương trình tough-up, tough-down đều cho phổ băng DNA được khuếch đại đa dạng hơn. Cụ thể, với mẫu trẻ khỏe mạnh, chương trình tough-up và tough-down có khuếch đại đoạn gen ngắn hơn 1,5 kb nằm liền kề với băng 1,5 kb trên điện di đồ, trong đó chương trình tough-up thể hiện rõ hơn chương trình tough-down. Ngoài ra, từ hệ gen vi khuẩn trẻ khỏe mạnh, chương trình tough-down, tough-up còn khuếch đại một băng DNA nằm ở vị trí băng 250 bp của DNA chuẩn nhưng băng này khá mờ, thể hiện là đoạn DNA không được khuếch đại từ vi khuẩn trội có trong mẫu. Ngược lại, ở trẻ tiêu chảy, chương trình đặc hiệu đã khuếch đại gen 16S rRNA đặc hiệu hơn so với băng tương ứng từ mẫu trẻ khỏe mạnh. DNA là một băng gọn, nét, không có băng phụ. Tuy nhiên, chương trình tough-up và tough-down cũng đều khuếch đại thêm 2 băng DNA có kích thước thấp hơn 1,5 kb và một băng sáng, nét, gọn, đặc hiệu có kích thước khoảng 250 bp (Hình 1). Trong đó, chương trình tough-up khuếch đại băng 250 bp sáng, nét hơn chương trình tough-down. Điều này có thể do ở những chu kỳ đầu, nhiệt độ gắn mồi giảm thấp giúp mồi bám vào đa dạng các sợi khuôn. Do đó, các chu trình đầu của chương trình PCR đã khuếch đại làm tăng mạnh nguồn gen này trong mẫu theo cấp số nhân tương tự như nghiên cứu của Pechgit và đồng tác giả [14]. Đoạn gen này có kích thước ngắn nên với chu kỳ kéo dài, chúng có khả năng tăng rất nhanh ở các chu kỳ sau do đó băng này được khuếch đại với số lượng lớn khi kiểm tra sản phẩm PCR trên gel điện di. Ở chương trình tough-down, băng này nhỏ hơn, mờ hơn (Hình 1). Nguyên nhân là do gen chỉ được khuếch đại ở những chu kỳ cuối khi nhiệt độ gắn mồi đã giảm thấp, nồng độ mồi đã giảm thấp. Kết quả này cũng tương tự như kết quả nghiên cứu của Moezi và đồng tác giả [15]. Kết quả trong Hình 1 đã khẳng định rằng chương trình PCR tough-up đã khuếch đại tốt và đa dạng sản phẩm PCR khi sử dụng cặp mồi 27F và 1527R.
Để xác định xem đoạn gen có kích thước khoảng 250 bp khuếch đại từ mẫu bệnh bằng chương trình tough-up từ hai đa hệ gen tách bằng phương pháp phenol:chloroform và kit GenJET có phải là gen 16S rRNA hay không, hai đoạn gen này được cắt khỏi gel, tinh chế lại và giải trình tự.
Kết quả giải trình tự cho thấy, cả hai gen có kích thước khoảng 250 bp được khuếch đại bằng chương trình tough-up đều có kích thước 247 bp, tương đồng giữa hai trình tự là 100% (Hình 2). Đặc biệt, trình tự có đầy đủ vị trí nhận biết của hai mồi, chứng tỏ đây là đoạn gen có mặt trong hệ gen, không phải các đoạn gen bị phân hủy, tự mồi với nhau. Kết quả này rất có giá trị để khẳng định sự ổn định và chất lượng của mẫu DNA metagenome sau khi tách chiết, đặc biệt là mẫu DNA được tách chiết từ phân của trẻ tiêu chảy có chứa nhiều tạp chất khác nhau. Sự phát hiện thấy cả hai mồi ở hai đầu đoạn gen đã gợi ra cho chúng tôi hai giả thuyết: (1) sự xuất hiện của vi khuẩn trong phân có gen 16S rRNA khác biệt với các nghiên cứu từ trước tới nay; (2) sự bắt cặp không đặc hiệu của mồi với các gen khác. Tuy nhiên, chúng tôi vẫn nghiêng về giả thuyết số một vì cho rằng gen 16S rRNA là gen ít bị biến đổi nhất và đặc thù nhất cho việc phân loại vi khuẩn. Để tìm hiểu xem đoạn gen đã được khuếch đại bằng cặp mồi này là gen gì, có phải là gen 16S rRNA không, chúng tôi tiến hành nghiên cứu phân tích, so sánh tương đồng của gen với gen trên ngân hàng gen.
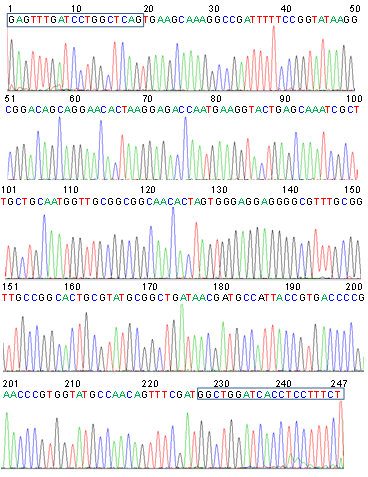
Hình 2. Sắc ký đồ giải trình tự sản phẩm PCR có kích thước khoảng 250 bp được khuếch đại bằng chương trình tough-up từ đa hệ gen của trẻ tiêu chảy không rõ nguyên nhân
Vùng đóng khung: trình tự mồi 27F và 1527R trên gen.
3.2. Phân tích trình tự gen 247 bp
Đoạn gen có kích thước 247 bp đã được so sánh trình tự gen với trình tự tương đồng trên ngân hàng gen NCBI. Kết quả (Hình 3) đã không như chúng tôi dự đoán, gen kích thước 247 bp tương đồng cao nhất (99,13%) với 22 gen mã hóa carbohydrate-binding protein (CBP), điển hình là gen có mã số CP04800. Ngoài ra, gen cũng tương đồng cao 98-99% với các gen khác như gen mã hóa bacterial Ig-like domain-containing protein (BIP), secreted protein with Ig (SPI), exo-beta-1,6-galactobiohydrolase (EBG) của các serovar trong loài B. longum (Hình 3). Mặc dù có các tên gọi khác nhau nhưng bản chất của các gen này đều là gen mã hóa cho CBP vì các enzyme thủy phân carbohydrate đã được chứng minh có chứa các vùng CBP, Ig-like giúp enzym bám tốt lên cơ chất [20]. Để tìm hiểu nguyên nhân cặp mồi 27F và 1527R lẽ ra chỉ bắt được với gen 16S RNA nhưng nay lại bắt không đặc hiệu với các gen nêu trên, chúng tôi đã đi tìm hiểu phân tích vùng mồi (vùng tô đậm trên Hình 3) có mức độ tương đồng với các gen được khuếch đại.

Hình 3. So sánh trình tự gen 247 nucleotide với các trình tự gen trên ngân hàng gen
Dãy chữ đứng trước trình tự gen chỉ mã số gen trên ngân hàng gen kèm theo viết tắt tên enzym mà gen mã hóa; CBP: carbohydrate-binding protein; BIP: bacterial Ig-like domain-containing protein; SPI: secreted protein with Ig; EBG: exo-beta-1,6-galactobiohydrolase. Vùng tô màu nâu là trình tự mồi. Các chữ được tô đậm thể hiện là các nucleotide tương đồng với nucleotide của mồi 27F và 1527R. Dấu * thể hiện vị trí có các nucleotide tương đồng trong các trình tự.
Kết quả phân tích vùng mồi cho thấy, ngoại trừ trình tự CBP trong hệ gen B. longum N2G10 C5930 mã số NJNV01000248 được giải trình tự bằng shotgun ra, các trình tự khác mã hóa các CBP nói trên đều có vùng trình tự tương đồng khá cao (13/20 nucleotide) với mồi 1527R và tập trung chủ yếu tại đầu 3’ của mồi (Hình 3) trong đó có ít nhất sáu nucleotide ở tận cùng đầu 3’ của mồi tương đồng 100% với trình tự trên gen, đặc biệt 5/6 nuleotide này thuộc loại purin nucleotide. Sự có mặt của các purin ở tận cùng đầu 3' giúp mồi bám được chắc với sợi khuôn.
Khác với mồi 1527R, mồi 27F chỉ tương đồng sáu đến bảy nucleotide ở tận cùng đầu 3’với các trình tự mã hóa cho các enzym nói trên trong đó đại diện là các trình tự được liệt kê trong Hình 3. Trong số 7 nucleotide này cũng có 5 nucleotide là purin. Purin là các nucleotide tạo 3 liên kết hydro với nucleotide trên sợi bổ sung làm cho mồi bám chặt với sợi khuôn hơn các nucleotide pirimidin.
Như vậy, trên cả hai mồi, sự có mặt của 5 nucleotide purin ở tận cùng đầu 3’ tương đồng với trình tự gen đích đã giúp mồi bám chắc với sợi khuôn để khuếch đại đoạn gen ở nhiệt độ thấp. Ở nhiệt độ nóng chảy cao hơn, số lượng liên kết hydro từ 5 nuleotide không đủ mạnh để hình thành liên kết với sợi khuôn nên mồi không bám được với sợi khuôn chặt, dễ bị tách ra khỏi sợi khuôn nên không khuếch đại được gen như trong trường hợp sử dụng chương trình đặc hiệu ở trên. Điều này nói lên tầm quan trọng của việc thiết kế mồi sử dụng các nucleotide purin ở đầu tận cùng 3’ sẽ giúp khả năng bám của mồi lên trình tự đích tốt và tăng cơ hội khuếch đại gen. Như vậy, gen có kích thước 247 bp được khuếch đại từ đa hệ gen vi khuẩn trong phân của bệnh nhi không phải là gen 16S rRNA mà là đoạn gen khác mã hóa cho vùng bám polysaccharide của enzym.
Để tìm hiểu liệu cặp mồi 27F và 1527R có khuếch đại được đầy đủ gen 16S rRNA của vi khuẩn Bifidobacterium có kích thước 1,5 kb không để đảm bảo khi sử dụng chương trình đặc hiệu, chúng tôi không bỏ sót chi vi khuẩn đóng vai trò rất quan trọng trong hệ tiêu hóa của người nói chung và của trẻ em nói riêng này, gen 16S rRNA của chủng chuẩn B. longum ATCC15708 mã số U10152 đã được chúng tôi sử dụng để tìm kiếm vùng tương đồng với trình tự của cặp mồi.
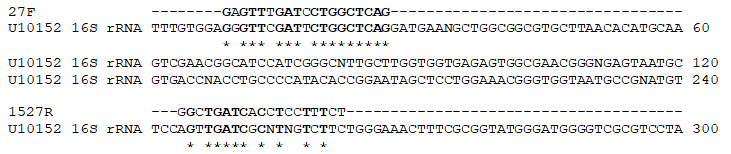
Hình 4. Vị trí tương đồng của mồi 27F và 1527R trên trình tự gen 16S rRNA của B. longum ATCC15708 mã số U10152 trên ngân hàng gen.
Dấu * thể hiện vị trí có các nucleotide tương đồng trong các trình tự; Dấu - chỉ không có trình tự tương đồng. Chữ đậm là các nucleotide tương đồng giữa mồi và gen.
Lại một lần nữa kết quả không như dự đoán, chúng tôi phát hiện ra gen 16S RNA của vi khuẩn longum ATCC15708 không thể khuếch đại được đầy đủ gen với kích thước 1,5 kb như những vi khuẩn khác vì mồi 1527R không tương đồng với vùng trình tự nào ở phần cuối gen. Thay vào đó, cặp mồi này may chăng có thể khuếch đại được vùng gen chỉ có kích thước khoảng 240 bp (Hình 4). Tuy nhiên, việc khuếch đại được đoạn gen này cũng vô cùng khó khăn do mồi 1527R không phù hợp. Quan sát trên Hình 4 có thể thấy mồi 27F cũng có sự tương đồng cao với trình tự gen đích: 16/19 nucleotide tương đồng, đặc biệt là chín nucleotide tận cùng đầu 3’ của mồi tương đồng 100% với trình tự trên gen đích. Ngược lại, trình tự mồi 1527R có 10/20 nucleotide tương đồng với trình tự trên gen 16S rRNA nhưng các nucleotide nằm rải rác trên trình tự và nằm cách đầu 5’ của gen khoảng 250 nucleotide (Hình 4). Đặc biệt nucleotide ở tận cùng đầu 3’ của mồi không bắt được với gen đích nên chắc chắn việc khuếch đại gen 16S rRNA bằng cặp mồi này từ hệ gen của Bifiodobacterium sẽ gặp khó khăn như trong nghiên cứu của Hayashi và đồng tác giả đã gặp phải [11]. Như vậy, nếu việc khuếch đại gen 16S rRNA của B. longum gặp khó khăn là do đoạn mồi 1527R, không phải do mồi 27F. Có thể đây là lý do mà trong nghiên cứu trước, thay vì sử dụng mồi 1527R, sử dụng mồi 35F, 529F và 1492R giúp cải thiện đáng kể khả năng khuếch đại đa dạng vi khuẩn Bifiodobacterium[11].
Gen 16S rRNA của vi khuẩn có kích thước 1,55 kb có cấu trúc gồm 9 vùng siêu biến (ký hiệu từ V1 đến V9) xen kẽ với các vùng bảo thủ (bảo tồn). Trong các nghiên cứu metagenomics đánh giá đa dạng quần thể vi khuẩn dựa trên gen 16S rRNA, thông thường các vùng siêu biến V3-V4, V3-V5, V6-V8 hoặc cả gen 16S rRNA sẽ được giải trình tự và phân tích. Mỗi vùng gen có ưu điểm và nhược điểm riêng và không có vùng nào có thể bao quát hết cả cộng đồng vi khuẩn trong hệ sinh thái. Điển hình vùng V1-V3 giúp phát hiện tốt các vi khuẩn thuộc chi Escherichia and Shigella [21]. Vùng V3 được đánh giá là vùng tốt nhất cho phân tích đa dạng vi khuẩn bằng PCR-DGGE [22], nhưng vùng gen này không có hiệu quả trong đánh giá đa dạng các vi khuẩn thuộc chi Clostridium và Staphylococcus. Hai chi vi khuẩn này lại được nhận diện tốt bằng vùng V6-V8 [23]. Việc phân tích các vùng gen siêu biến sẽ ít có cơ hội phân loại được vi khuẩn đến loài nhưng có khả năng làm tăng tính đa dạng của vi khuẩn. Ngược lại, việc sử dụng toàn bộ gen 16S rRNA giúp phân loại vi khuẩn được chính xác, tăng cơ hội định danh được đến loài nhưng cũng bị hạn chế ở các vi khuẩn có trình gen khác biệt so với mồi sử dụng. Vì vậy, tùy theo mục đích nghiên cứu và đặc điểm hệ vi khuẩn mà các nhà nghiên cứu có thể lựa chọn các vùng gen, các phương pháp đánh giá đa dạng cho phù hợp.
Tóm lại, nghiên cứu này đã chỉ ra rằng cặp mồi 27F, 1527R không tương đồng cao với gen 16S rRNA của vi khuẩn chi Bifidobacterium nên khó được áp dụng để đánh giá đa dạng hệ vi khuẩn bằng phương pháp giải trình tự metagenom vi khuẩn ở các môi trường có Bifidobacterium chiếm ưu thế. Cặp mồi này cũng khó được áp dụng để phân loại vi khuẩn trong chi Bifidobacterium. Việc giảm nhiệt độ gắn mồi giúp tăng cơ hội bám của mồi 27F nhưng dẫn đến việc khuếch đại đoạn gen không đặc hiệu của Bifidobacterium. Phát hiện mới trong nghiên cứu này đã mở ra ứng dụng của cặp mồi cho việc phát hiện sơ bộ sự có mặt của chi Bifidobacterium trong môi trường sinh thái, phân biệt với các loại vi khuẩn khác. Tuy nhiên, với sự hạn chế về số lượng mẫu, phát hiện mới này cũng cần được khẳng định khi thực hiện trên cỡ mẫu lớn hơn. Ngoài ra, nghiên cứu này cũng khẳng định cặp mồi 27F, 1527R là cặp mồi hiệu quả để: (1) phân loại vi khuẩn không thuộc chi Bifidobacterium; (2) đánh giá đa dạng vi khuẩn trong hệ sinh thái dựa trên quan sát sự phân ly vùng gen được khuếch đại bằng chương trình tough-up trên điện di đồ DGGE hay RFLP đồng thời cặp mồi vẫn có thể sử dụng để so sánh sự đa dạng của hệ vi sinh vật trong các mẫu tương đồng nhau thông qua phổ băng DNA (dấu vân tay phân tử) khi sử dụng chương trình tough-up.
4. KẾT LUẬN
Sử dụng chương trình tough-up PCR với nhiệt độ gắn mồi ban đầu 50oC đã làm cho mồi 27F, 1527R khuếch đại đoạn gen 247 bp không phải gen 16S rRNA từ vi khuẩn Bifidobacterium. Với nhiệt độ gắn mồi thấp, các mồi có trình tự purin đầu 3’ tương đồng với sợi khuôn 5-6 nucleotide có khả năng bám tốt với sợi khuôn. Nhiệt độ gắn mồi cao làm cho việc khuếch đại gen đặc hiệu.
Lời cảm ơn: Công trình được hỗ trợ kinh phí từ đề tài nghiên cứu mã số ĐTĐLCN.63/22 (Bộ Khoa học và Công nghệ) và trang thiết bị của Phòng Thí nghiệm trọng điểm Công nghệ gen tại Viện Công nghệ sinh học, Viện Hàn lâm Khoa học và Công nghệ Việt Nam (VAST).
Tài liệu tham khảo
2. Pukall R., Päuker O., Buntefuß D., Ulrichs G., Lebaron P., Bernard L., Guindulain T., Vives-Rego J., Stackebrandt E., High sequence diversity of Alteromonas macleodii-related cloned and cellular 16S rDNAs from a Mediterranean seawater mesocosm experiment, FEMS Microbiology Ecology, 1999, 28(4):335-344. DOI: 10.1111/j.1574-6941.1999.tb00588.x
3. Phan Thùy Dương, Lê Phụng Hiển, Nguyễn Thị Xuân, Trần Thị Thanh Huyền, Mai Thị Đàm Linh, Phạm Thanh Hiền, Nguyễn Thị Vân Anh, Bùi Thị Việt Hà, Đặc tính probiotic của chủng Lactobacillus gasseri SMH15 được phân lập từ sữa mẹ, Tạp chí Công thương, 2020, https://tapchicongthuong.vn/dac-tinh-probiotic-cua-chung-lactobacillus-gasseri-smh15-duoc-phan-lap-tu-sua-me-72743.htm.
4. Yu Z., Zhou N., Qiao H., Qiu J., Identification, cloning, and expression of L-amino acid oxidase from marine Pseudoalteromonas sp. B3, The Scientific World Journal, 2014, 1:979858. DOI:10.1155/2014/979858
5. Westaway J. A. F., Huerlimann R., Miller C. M., Kandasamy Y., Norton R., Rudd D., Methods for exploring the faecal microbiome of premature infants: a review, Maternal Health, Neonatology and Perinatology, 2021, 7:11. DOI: 10.1186/s40748-021-00131-9
6. Rathsack K., Böllmann J., Martienssen M., Comparative study of different methods for analyzing denitrifying bacteria in fresh water ecosystems, Journal of Water Resource and Protection, 2014, 6(6):609-617. DOI: 10.4236/jwarp.2014.66059
7. Pukall R., Päuker O., Buntefuß D., Ulrichs G., Lebaron P., Bernard L., Guindulain T., Vives-Rego J. Stackebrandt, E., High sequence diversity of Alteromonas macleodii-related cloned and cellular 16S rDNAs from a Mediterranean seawater mesocosm experiment, FEMS Microbiology Ecology, 1999, 28(4):335-344. DOI: 10.1111/j.1574-6941.1999.tb00588.x
8. Ramírez M., López-Piñeiro A., Peña D., Rato Nunes J., Albarrán Á., Muñoz A., Gama J., Loures L., Seasonal and interannual fuctuation of the microbial soil community in a maize field under long-term conservation agriculture management, Sustainability, 2017, 9(5):778. DOI: 10.3390/su9050778
9. Nitipan S., Mamimin C., Intrasungkha N., Birkeland N. K., O-Thong S., Microbial community analysis of thermophilic mixed culture sludge for biohydrogen production from palm oil mill effluent, International Journal of Hydrogen Energy, 2014, 39(33):19285-19293. DOI: 10.1016/j.ijhydene.2014.05.139
10. Sipos R., Székely A. J., Palatinszky M., Révész S., Márialigeti K., Nikolausz M., Effect of primer mismatch, annealing temperature and PCR cycle number on 16S rRNA gene-targetting bacterial community analysis, FEMS Microbiology Ecology, 2007, 60(2):341-350. DOI: 10.1111/j.1574-6941.2007.00283.x
11. Hayashi H., Sakamoto M., Benno Y., Evaluation of three different forward primers by terminal restriction fragment length polymorphism analysis for determination of fecal bifidobacterium spp. in healthy subjects, Microbiology and Immunology, 2004, 48(1):1-6. DOI: 10.1111/j.1348-0421.2004.tb03481.x
12. Borah P., Primer designing for PCR, Science Vision, 2011, 11(3):134-136.
13. Ishii K., Fukui M., Optimization of annealing temperature to reduce bias caused by a primer mismatch in multitemplate PCR, Applied and Environmental Microbiology, 2001, 67(8):3753-3755. DOI: 10.1128/AEM.67.8.3753-3755.2001
14. Pechgit P., Intarapuk A., Pinyoowong D., Bhumiratana A., Touchdown-touchup nested PCR for low-copy gene detection of benzimidazole-susceptible Wuchereria bancrofti with a Wolbachia endosymbiont imported by migrant carriers, Experimental Parasitology, 2011, 127(2): 559-568. DOI: 10.1016/j.exppara.2010.10.022
15. Moezi P., Kargar M., Doosti A., Khoshneviszadeh M., Multiplex touchdown PCR assay to enhance specificity and sensitivity for concurrent detection of four foodborne pathogens in raw milk, Journal of Applied Microbiology, 2019, 127(1):262-273. DOI: 10.1111/jam.14285
16. Vũ Thị Thu Hà, Nguyễn Tuấn Khiêm, Tăng Chí Thượng, Trần Thị Mộng Hiệp, Khảo sát đặc điểm bệnh tiêu chảy kéo dài ở trẻ em tại bệnh viện Nhi đồng 2, Nghiên cứu Y học - Y học TP Hồ Chí Minh, 2016, 20(2):96-102.
17. Kieser S., Sarker S. A., Sakwinska O., Foata F., Sultana S., Khan Z., Islam S., Porta N., Combremont S., Betrisey B., Fournier C., Charpagne A., Descombes P., Mercenier A., Berger B., Brüssow H., Bangladeshi children with acute diarrhoea show faecal microbiomes with increased Streptococcus abundance, irrespective of diarrhoea aetiology, Environmental Microbiology, 2018, 20(6):2256-2269. DOI: 10.1111/1462-2920.14274
18. Mai V., Braden C. R., Heckendorf J., Pironis B., Hirshon J. M., Monitoring of stool microbiota in subjects with diarrhea indicates distortions in composition, Journal of Clinical Microbiology, 2006, 44(12):4550-4552. DOI: 10.1128/JCM.01542-06
19. Nguyen T. Q., Phung T. B. T., Ngoc T. T., Do T. H., A comparison of five methods for effective extraction of bacterial metagenomic DNA from stools of children suffer from diarrhea with unknown cause, Biological Forum - An International Journal, 2023, 15(8a):1-9.
20. Nguyen K. H. V., Dao T. K., Nguyen H. D., Nguyen K. H., Nguyen T. Q., Nguyen T. T., Nguyen T. M. P., Truong N. H., Do T. H., Some characters of bacterial cellulases in goats’ rumen elucidated by metagenomic DNA analysis and the role of fibronectin 3 module for endoglucanase function, Animal Bioscience, 2021, 34(5):867-879. DOI: 10.5713/ajas.20.0115
21. Johnson J. S., Spakowicz D. J., Hong B.Y., Petersen L. M., Demkowicz P., Chen L., Leopold S. R., Hanson B. M., Agresta H. O., Gerstein M., Sodergren E., Weinstock G. M., Evaluation of 16S rRNA gene sequencing for species and strain-level microbiome analysis, Nature Communications, 2019, 10(1):5029. DOI: 10.1038/s41467-019-13036-1
22. Yu Z., Morrison M., Comparisons of different hypervariable regions of rrs genes for use in fingerprinting of microbial communities by PCR-denaturing gradient gel electrophoresis, Applied and Environmental Microbiology, 2004, 70(8):4800-4806. DOI: 10.1128/AEM.70.8.4800-4806.2004
23. Yang B., Wang Y., Qian P.Y., Sensitivity and correlation of hypervariable regions in 16S rRNA genes in phylogenetic analysis, BMC bioinformatics, 2016, 17(1):135. DOI: 10.1186/s12859-016-0992-y